近日,信息工程学院余振华副教授课题组在单细胞点突变数据的聚类研究中取得新进展。单细胞点突变数据聚类对于揭示肿瘤内部的亚克隆及其进化关系有着重要的研究意义,有助于重构癌症发展历史并辅助临床设计有效的个性化治疗方案。由于单细胞数据中存在假阳性、假阴性、数据缺失等复杂问题,而假阴性率可达80%以上,同时数据缺失率高达50%,因此如何高效准确地对数据进行聚类是当前单细胞测序领域的一个重要研究方向。
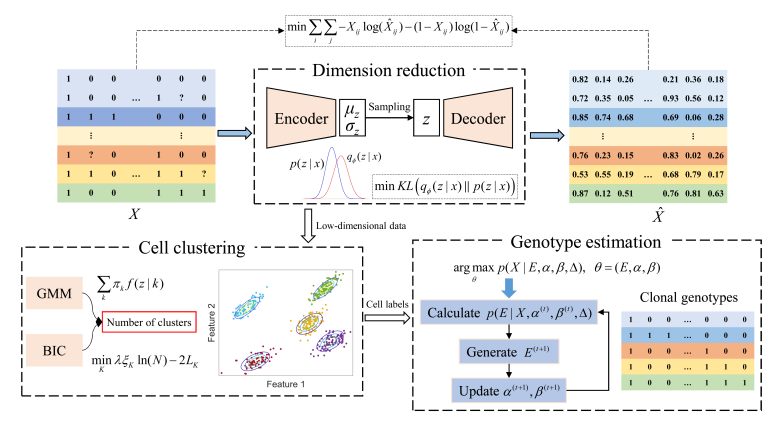
为解决这一难题,余振华课题组提出了一种基于无监督表征学习的计算方法,在隐含低维空间中对细胞进行表征和聚类,并结合传统的MCMC、EM等方法对细胞基因型进行准确估计,增强了预测结果的可解释性。这一成果对于揭示肿瘤发生和发展的生物学机理具有重要推动作用。该成果以“bmVAE: a variational autoencoder method for clustering single-cell mutation data”为题被生物信息学顶级期刊《Bioinformatics》录用,论文第一作者为我院计算机技术在读硕士研究生延佳倩,通讯作者为我院余振华副教授。
本研究工作获得国家自然科学基金青年科学基金项目、宁夏回族自治区青年科技人才托举工程和中科院“西部之光”青年学者等项目的资助。
